青岛


近日,厦门大学海洋与地球学院陈仕玺教授课题组与生命科学学院郭峰副教授课题组在Microbiome期刊在线发表了题为“Robust host source tracking building on the divergent and non-stochastic assembly of gut microbiomes in wild and farmed large yellow croaker”的研究论文,探究了野生与养殖大黄鱼的肠道微生物的组成与功能上的差异,并利用上述特征建立了可靠的大黄鱼溯源技术方案。
海水鱼类的溯源技术,暨如何确定采集的海水鱼是源自野生或养殖环境,不仅影响着价格和食品安全,也有助于野生资源量的合理评估。目前,全球许多重要海水养殖鱼类尚未完全建立可用于区分养殖和野生个体的遗传分子标记。肠道菌群的组成和功能与宿主及其栖息的环境存在关联。因此,通过分析道菌群的组成与功能上的差异,可对人类等陆生动物进行溯源分析,但相关研究在鱼类中还鲜有报道。
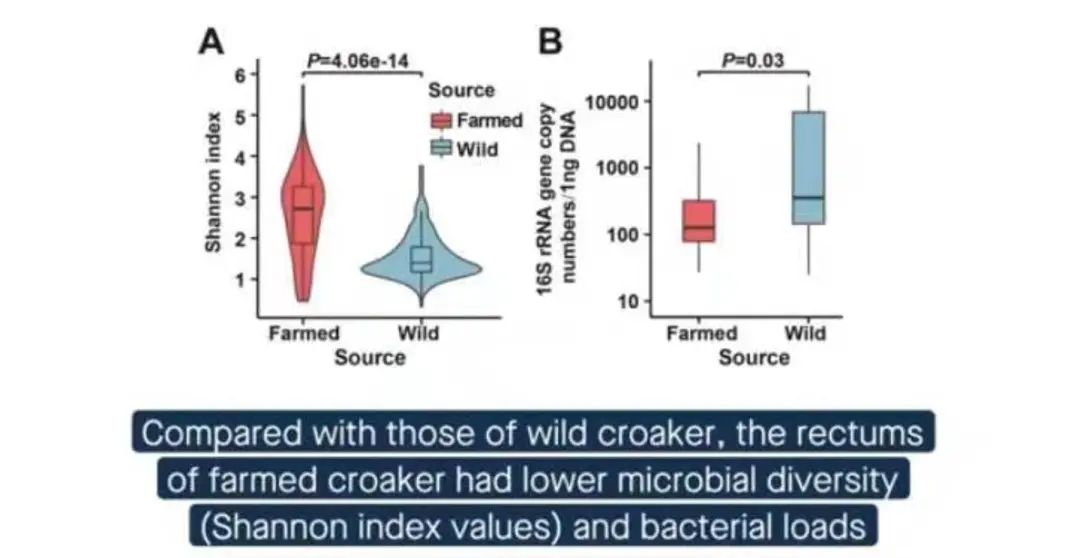
大黄鱼是我国最为重要的海水经济鱼类,曾经为四大海产之一,素有“国鱼”之称。但由于过度捕捞,大黄鱼的野生种群资源枯竭。经过水产科技工作者的不懈努力,目前大黄鱼的养殖规模位居海水养殖鱼类之首。该研究采集了10批次的共计291尾大黄鱼样品(野生212尾,养殖79尾),利用高通量测序和生物信息学手段分析了野生与养殖大黄鱼的肠道微生物群落结构。数据分析表明,野生和养殖大黄鱼的肠道微生物在群落结构具有明显的差异,针对分离代表菌株和菌群水平的功能预测提示,部分群落结构差异与食物有关。同时,研究发现来自野生和养殖大黄鱼的肠道菌群差异并非“非此即彼”,聚类和降维分析显示二者存在一定重叠,并且不同批次间的个体的差异同样显著,符合肉食性鱼类肠道菌群特点,提示菌群组成可能具有随机性。为了排除肠道菌群的群落组装是随机过程占主导因素,本研究采用生态学中性模型以及BetaNTI参数对菌群进行检验,结果表明不论是养殖或野生大黄鱼的肠道菌群,非随机过程与随机过程对群落组装的影响程度相当或更高。在此基础上,本研究进一步采用优化的随机森林预测算法,实现了对未知个体(包括新的样本批次)的可靠溯源,准确率可达93%。
该研究首次基于肠道菌群实现了海水肉食鱼类的溯源,并阐释了群落组装模式与可靠溯源之间的联系,为采用类似方法开展宿主溯源的研究提供重要参考,也为包括大黄鱼等海洋经济鱼类的来源溯源提供了新的思路。
厦门大学生命科学学院硕士生朱俊、博士生李浩和厦门大学海洋与地球学院硕士生景泽周为本文共同第一作者,郭峰副教授和陈仕玺教授为该论文的共同通讯作者。该论文得到了国家重点研发计划(No. 2018YFC1406303)、国家自然科学基金(No. 31670492 and No. 41976092),南方海洋科学与工程广东省实验室(珠海)创新团队项目(No. 311021006)和中央高校基本科研业务费专项资金(No. 20720200114)的资助。
论文链接:https://doi.org/10.1186/s40168-021-01214-7(链接中含有视频摘要)
本文来源:厦门大学