中国科学院青岛生物能源与过程研究所基于基因组靶向挖掘发现真菌黄酮生物合成新机制
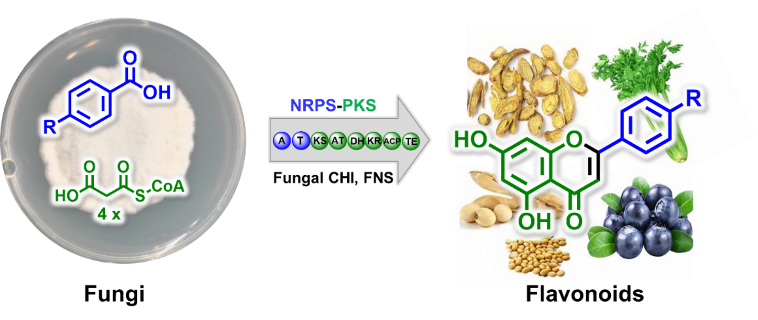
近日,中国科学院青岛生物能源与过程研究所吕雪峰团队在丝状真菌中发现了黄酮生物合成基因簇(BGCs),通过合成途径解析阐明了真菌黄酮合成新机制,丰富了对黄酮类化合物生物合成的认识,同时为合成生物技术开发提供了新的选择(图1),相关研究成果发表在《德国应用化学》。黄酮是广泛存在于植物中的一大类天然产物,如花青素、大豆异黄酮、水飞蓟宾等,在功能性食品、医药等领域具有重要的应用价值。目前,黄酮资源依赖于植物中获得,受制于植物种植周期长、组分多含量低、分离纯化工艺复杂等诸多弊端,产能小、成本高,严重限制了黄酮类产品的应用开发和市场拓展。利用合成生物技术设计构建黄酮细胞工厂,推动植物黄酮的微生物高效生产成为一种重要解决方案。由于物种差异,植物黄酮合成途径在微生物中异源重构面临着适配性差、产量低等问题,距离商业化仍有较大差距。
图1. 真菌黄酮合成示意图
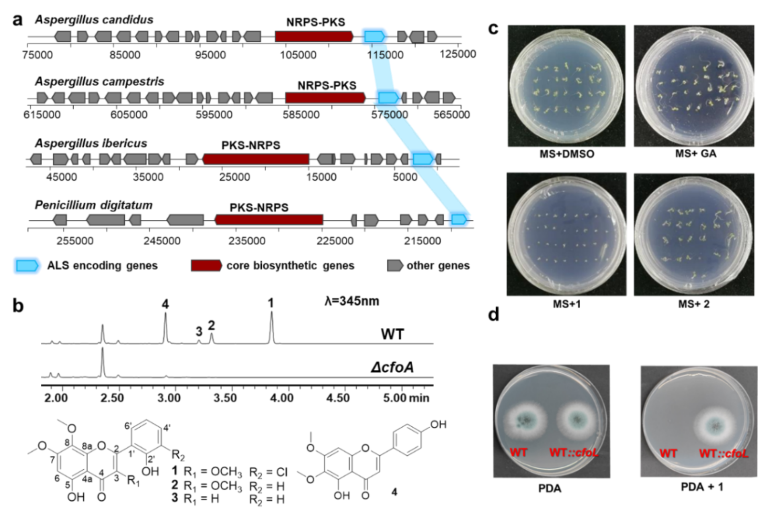
利用自抗性基因共定位(SRGD)策略是开展天然产物基因组挖掘的一种有效方式,为了寻找具有抗菌和除草活性的天然产物,研究团队首先以合成支链氨基酸的关键酶乙酰乳酸合酶(ALS)为探针,在亮白曲霉中发现了负责合成黄酮类化合物氯黄酮的生物合成基因簇(图2)。活性评价显示氯黄酮具有抑制拟南芥种子萌发和抑制病原菌生长的活性,而体内和体外实验也证实cfoL确实是编码ALS的氯黄酮自抗性基因,因此具有开发成为除草剂和抗生素的潜力。
图2. SRGD策略发现真菌氯黄酮合成基因簇及活性分析
植物黄酮的生物合成途径目前已经研究清楚,首先是查耳酮合酶(CHS)III型PKS以对羟基肉桂酰辅酶A为起始单元与3分子乙酰辅酶A缩合形成查耳酮;然后在查耳酮异构酶(CHI)作用下形成黄烷酮,由黄酮合酶(FNS)进一步催化黄烷酮形成黄酮骨架,最后在修饰酶的作用下形成多种多样的黄酮类化合物。本研究中通过基因敲除、同位素标记实验发现亮白曲霉中查尔酮骨架由NRPS-PKS杂合酶CfoA以苯甲酸或对羟基苯甲酸为起始单元与4分子乙酰辅酶A缩合形成,而非是植物的III型PKS装配模式,说明真菌黄酮骨架合成机制与植物存在显著差异。
图3. 亮白曲霉氯黄酮生物合成途径
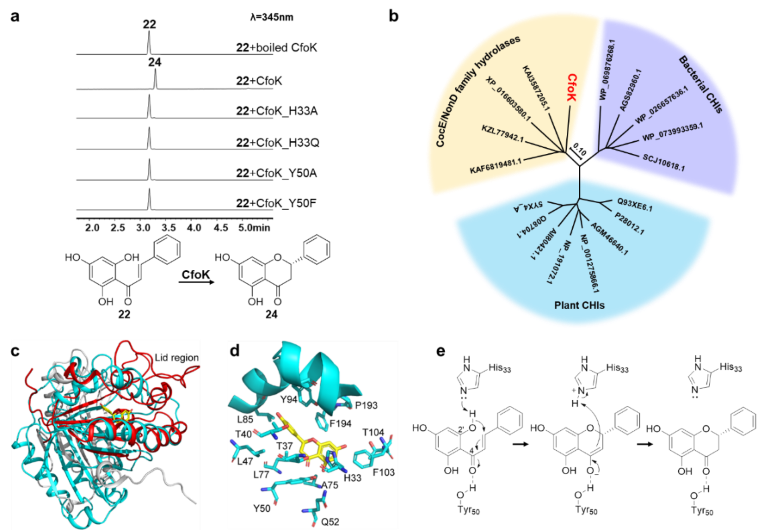
在此基础上,通过基因敲除和体外酶活实验发现CfoK催化查耳酮关环生成黄烷酮,这与植物中的CHI具有相同的功能。但是CfoK与目前已经发现的CHI存在不同的进化关系。基于AlphaFold2预测的蛋白质结构模拟和定点突变实验表明CfoK通过His33介导的酸碱催化诱发查耳酮发生Oxa-Michael加成反应,以6-endo-trig关环方式形成构型专一的黄烷酮(图4),这一机制不同于植物CHI中水介导的催化过程,因此CfoK是一种新颖的真菌查耳酮异构酶。
图4. 真菌中查耳酮异构酶CfoK的表征
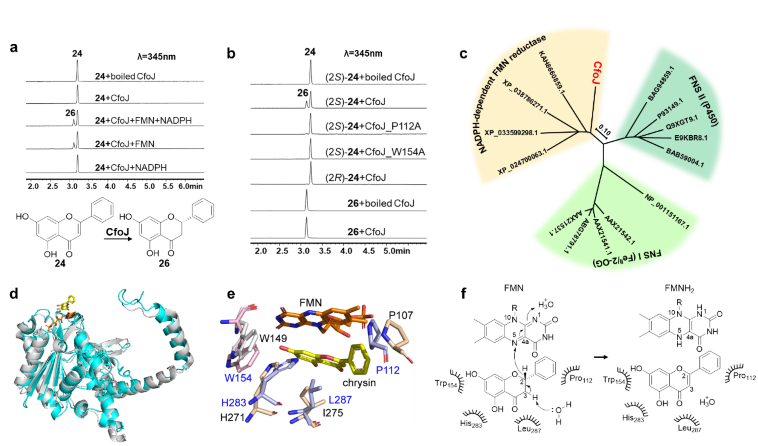
亮白曲霉中由黄素依赖型氧化还原酶CfoJ行使黄酮合酶(FNS)功能,将黄烷酮转化为黄酮,进化树分析显示其与已知的FNS I (2-酮戊二酸依赖型双加氧酶)和FNS II(细胞色素氧化酶P450)处于不同的分支。蛋白质结构预测和定点突变实验结果显示CfoJ是通过典型的黄素依赖型氧化还原酶催化机制介导C2-C3位的脱氢,将黄烷酮转化为黄酮,这与植物FNS的自由基催化机制完全不同,因此CfoJ是一种新型的真菌黄酮合酶(图5)。
图5. 真菌中黄酮合酶CfoJ的表征
该研究完整解析了真菌黄酮独特的生物合成途径,提示真菌黄酮合成途径在进化上是独立的,而非通过基因水平转移从植物中获得。这一发现丰富了自然界黄酮生物合成的多样性,同时也为其合成生物技术开发与微生物高效制造提供了新思路,因此对黄酮的应用价值开发具有重要意义。微生物制造工程中心吕雪峰研究员和黄雪年研究员为本论文的共同通讯作者,博士研究生张伟为论文的第一作者,能源作物分子育种团队付春祥研究员和吴振映副研究员参与了此项工作。本研究获得了国家重点研发计划、国家自然科学基金、山东省人才计划及山东省重点研发计划的支持。
原文链接: https://onlinelibrary.wiley.com/doi/abs/10.1002/anie.202215529
本文来源:中国科学院青岛生物能源与过程研究所