青岛


近日,山东大学海洋学院杜宗军教授团队解析了不同海洋大藻的核心微生物类群,发现了其强大的多糖降解潜力和次级代谢产物合成潜力,并对藻际微生物区系和周围环境微生物区系进行了比较,研究成果以“Epiphytic common core bacteria in the microbiomes of co-located green (Ulva), brown (Saccharina) and red (Grateloupia, Gelidium) macroalgae”为题发表在Microbiome。山东大学海洋学院教授杜宗军和德国马克斯普朗克研究所海洋微生物研究所博士Hanno Teeling为该论文共同通讯作者,两单位联合培养博士生卢德臣为本论文第一作者,马克斯普朗克研究所教授Rudolf I Amann、博士生王凤青为共同作者,山东大学为第一完成单位。
藻际微生物是一个十分有趣的研究课题,从生态以及技术应用方面都值得深入研究。由于对常见大型藻类附生细菌的多样性和生态生理作用普遍缺乏了解,目前尚不清楚哪些因素决定大型藻附生微生物菌落的初始定植以及定植后维持稳定的机制,大藻附生微生物的开发潜力也有待深入探究。
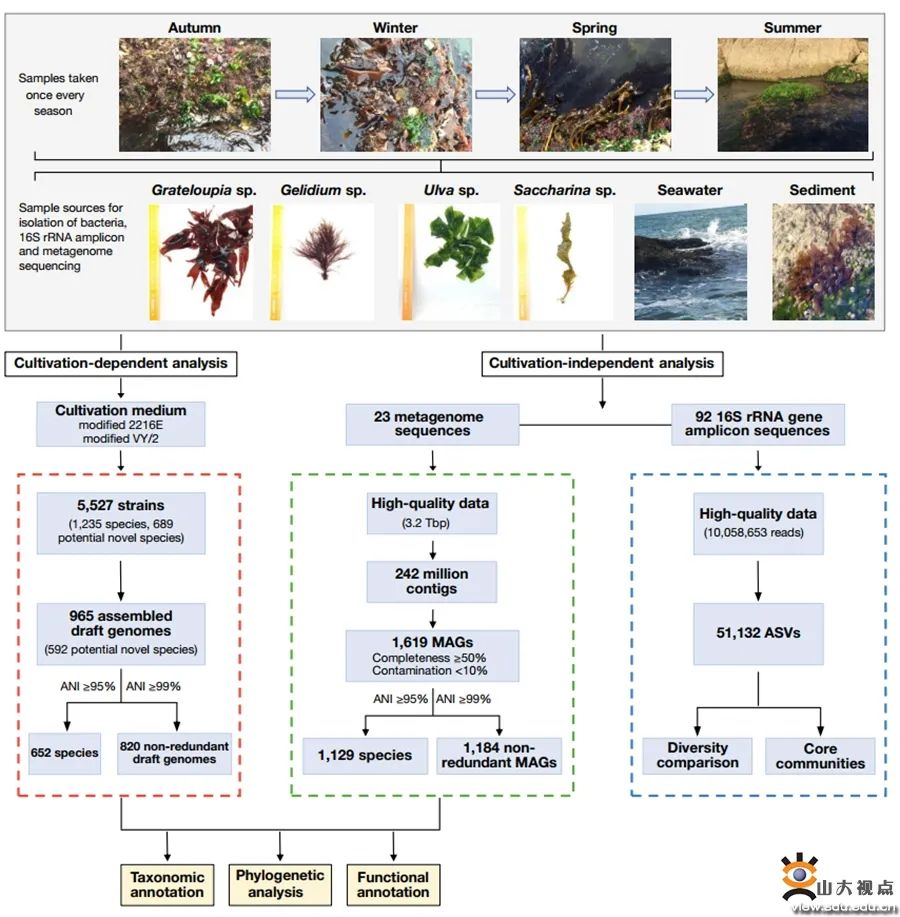
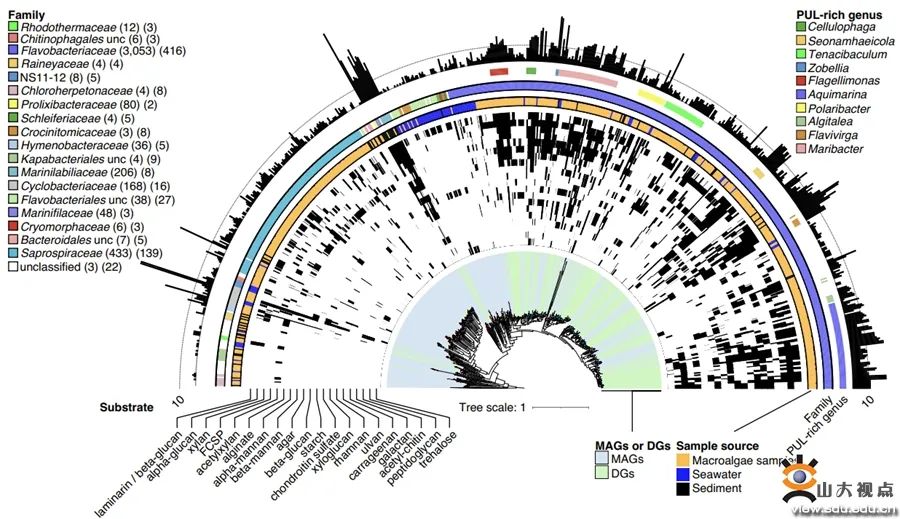
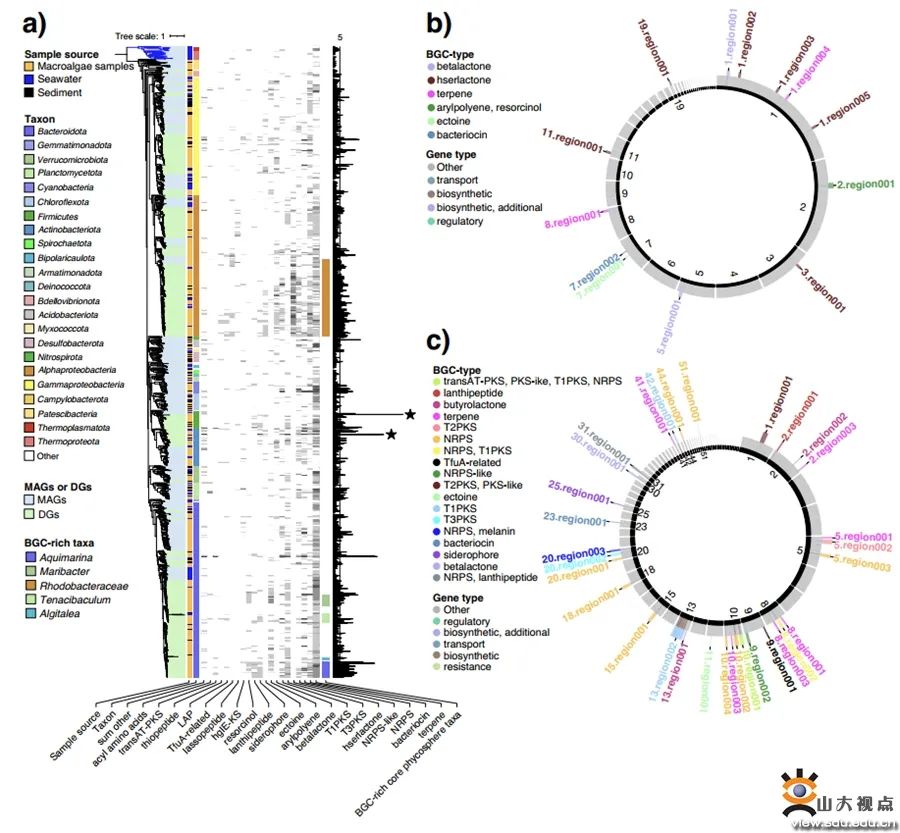
本研究对三个门、四种不同大型藻类的附生细菌群落进行了广泛而深入的研究。对中国威海近海礁石上的四类大藻进行了四个季节的样品采集,并以海水和沉积物作为对照组。通过16S rRNA扩增子测序、宏基因组分析、大规模细菌分离培养,纯培养菌株的基因组测序,结合细菌培养、生理学、分类学、基因组学、宏基因组学和生物信息学分析方面的专业知识,形成了目前最全面和详细的藻际细菌数据集。这项研究得到大量新的藻际微生物菌种资源以及大量藻际微生物的基因组草图和MAG,可作为未来生物技术勘探的候选菌株与材料。本研究鉴定了藻际微生物14个核心属(持续稳定的存在于所有大型藻类中)和14个优势属(持续稳定的存在于四种大型藻类中的三种)。针对藻类附生细菌的两个关键代谢特征:藻类多糖降解和生物活性次生代谢物合成潜力的研究发现,核心/优势细菌中含有十分丰富且多样的相关代谢途径。研究还发现藻际微生物在多糖降解潜力方面存在相当大的功能冗余,这可能代表细菌在不同藻类宿主定殖的一种适应性机制。次级代谢产物的研究中发现藻际微生物特别是黄杆菌,作为生物活性底物(> 8,000个生物合成基因簇)的来源可能被低估,并且作者发现红杆菌科可能通过高丝氨酸内酯控制和维持藻类定殖。这项研究极大地增进了我们对大型藻类附生细菌群落及其功能的了解和认知。
图一:实验及分析流程图
图二:MAG和纯培养细菌基因组中的PUL种类与数量分布
图三:a)1,619个MAG和965个基因组草图(DG)中的生物合成基因簇种类组成和分布,b)富含高丝氨酸内酯的基因组展示,c)基因簇数量最多的基因组展示
本文来源:山东大学