
北京大学等研究揭示蓝藻生物钟蛋白KaiC单体与单体间的协同相互作用
近日,北京大学物理学院凝聚态物理与材料物理研究所/定量生物学中心欧阳颀教授课题组及其合作者在Nature Communications在线发表题为“Determining subunit-subunit interaction from statistics of cryo-EM images: observation of nearest-neighbor coupling in a circadian clock protein complex”的研究论文。该研究基于单颗粒冷冻电镜技术解析了蓝藻生物钟蛋白KaiC关键突变体的高分辨结构,并利用统计物理模型揭示了KaiC六聚体的单体之间存在协同相互作用,为蓝藻生物钟实现稳定振荡的分子机制提供了直接证据。
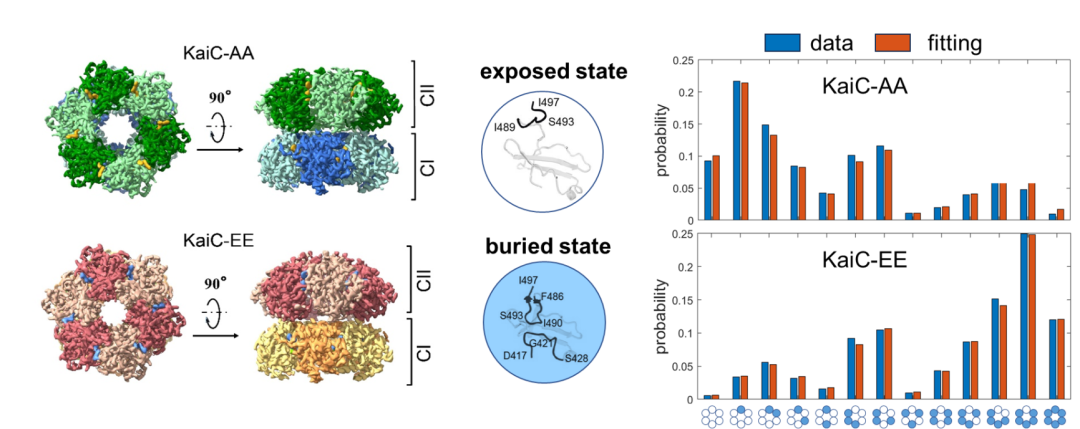
左图为KaiC-AA与KaiC-EE的电子密度示意图;中图为A-loop的两种构象;右图为KaiC-AA与KaiC-EE六聚体的A-loop构象分布的实验值和Ising模型的拟合值
蓝藻生物钟的核心部分由KaiA、KaiB、KaiC三个蛋白质构成,在KaiA与KaiB的辅助下,KaiC的磷酸化程度可在体外能实现周期近24小时的振荡。以往的结构研究解析了KaiC不同磷酸化状态突变体的晶体结构,但在不同突变体的晶体结构之间并没有观测到与磷酸化状态相关的结构差异。该研究利用冷冻电镜对模拟完全去磷酸化的突变体KaiC-AA以及模拟完全磷酸化的突变体KaiC-EE进行结构解析,得到了两种突变体的高分辨六聚体结构。研究基于高分辨结构,对KaiC单体的构象做进一步分类发现KaiC单体的A-loop片段存在2个主要构象:埋藏态和暴露态。在埋藏态中,KaiC的A-loop片段具有稳定的结构,而在暴露态中,A-loop片段无固定结构。基于单体的构象的统计数据,该研究建立了这两中突变体的能量景观,发现六聚体的构象分布可用统计物理中的Ising模型进行定量拟合。该结果说明KaiC六聚体的单体之间存在协同相互作用,且六聚体的构象分布受磷酸化状态的调控。在一个动力学系统中,稳定的振荡行为需要系统中存在非线性负反馈。该研究发现单体之间存在协同相互作用,为KaiC蛋白质构象与磷酸化状态之间的非线性反馈提供了直接观测证据。该工作结合冷冻电镜技术与统计物理的研究方法,对从冷冻电镜数据中挖掘蛋白质的构象分布信息进行统计物理分析,提出了冷冻电镜数据结合统计物理的研究方法解析蛋白质机器工作原理的新的思路。美国芝加哥大学Michael Rust教授、美国IBM沃森研究中心的涂豫海教授和欧阳颀为共同通讯作者。北京大学物理学院2017级博士研究生韩旭为该论文的第一作者。该工作得到国家自然科学基金委等机构的资助。
论文链接:
https://www.nature.com/articles/s41467-023-41575-1
本文来源:北京大学



